Rastrear matéria-prima é alternativa para auxiliar no controle da produção sucroenergética
Por Bernardo de Castro, presidente da divisão de Agricultura da Hexagon
A cultura soja se tornou a cadeia agrícola de maior importância para a economia brasileira (MAPA, 2017), possuindo pragas e doenças que cada vez mais ganham resistência aos métodos de controle químico utilizados por seu uso indiscriminado (Oliveira et al., 2016).
A partir da descoberta de moléculas que atuam silenciando genes específicos, sendo nomeado como RNA de interferência (RNAi), vários métodos de utilização e aplicação foram e estão sendo desenvolvidos no intuito de revolucionar métodos de controle de diversos organismos (Hannon, 2002; Boutros & Ahringer, 2008).
O mecanismo de RNA interferente (RNAi) é um processo que ocorre naturalmente em organismos eucarióticos. Trata-se de um processo que causa o silenciamento de genes antes de sua tradução, pela degradação do mRNA (Obbard, et al., 2009). Este processo foi descrito primeiramente em plantas e nomeado como silenciamento gênico pós-transcricional, ou PTGS (Jorgensen, et al., 1996).
O RNAi apresenta grande potencial biotecnológico devido a sua elevada especificidade, podendo assim colaborar para o campo da entomologia (Gordon & Waterhouse, 2007), seja para o estudo funcional de genes ou mesmo como uma ferramenta alternativa para o controle de insetos praga na agricultura (Price & Gatehouse, 2008). De maneira geral, estudos de RNAi nesses insetos é realizado através da utilização de plantas geneticamente modificadas para expressarem RNAs de dupla fita (dsRNAs) ou utilizando moléculas exógenas introduzidas por meio de microinjeção (Fishilevich et al., 2016).
Este trabalho tem como objetivo avaliar a viabilidade da utilização do mecanismo de RNAi para o estudo de genes em insetos, por via alimentar, em vagens de soja. Essa metodologia, conhecida como “Aplicação Ectópica”, se baseia na absorção direta do dsRNA no tecido vegetal, sendo posteriormente absorvida pelo inseto após se alimentar das plantas, resultando no silenciamento gênico. Trata-se de uma metodologia menos onerosa quando comparada às utilizadas tradicionalmente, que já vem sendo empregado com sucesso em estudos em outros organismos e alguns insetos. Neste trabalho, foi avaliada a capacidade de translocação do dsRNA em vagens de soja. Para isso, foram utilizadas moléculas de dsRNA sem complementaridade com os transcritos da soja.
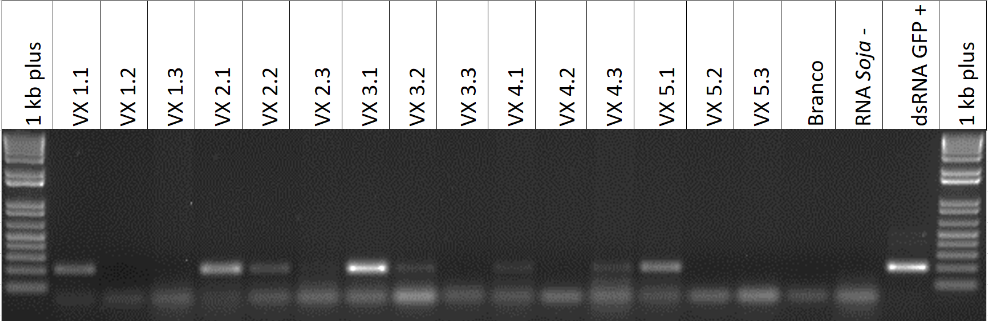
O experimento foi conduzido no laboratório de biotecnologia da Embrapa Soja, em Londrina, PR, durante o ano de 2017. Para avaliar a capacidade de translocação da solução de dsRNA nas vagens, foram preparados 5 tubos de 2 mL com uma microgota no fundo dos mesmos contendo 50 µL de [FC1] solução com concentração de 10 ng de dsRNA GFP. Após foram coletadas 5 vagens de soja BRS 1001 BTRR padronizadas com 3 sementes. Previamente a introdução da vagem no tubo, foi feito um corte de parte do pedúnculo para manter a parte viva em contato com a solução. Posteriormente o pedúnculo da vagem foi colocado em contato na microgota no [FC2] interior do tubo e armazenado por 24h em BOD à 26°C com fotoperíodo de 14h sendo o volume totalmente absorvido neste período de tempo. Após, as vagens foram retiradas dos tubos, lavadas com água destilada, seccionadas em três partes referentes aos segmentos das sementes, separadas as valvas das sementes e congeladas em nitrogênio líquido. Posteriormente foram colocadas em tubos de 2 mL para identificação e armazenamento em freezer à -80°C. As valvas foram separadas e identificadas da seguinte maneira: VX 1.1, 1.2, 1.3; VX 2.1, 2.2 e 2.3; VX 3.1, 3.2 e 3.3; VX 4.1, 4.2 e 4.3 e VX 5.1, 5.2 e 5.3 sendo referente à repetição da vagem após a secção.
O RNA total de cada secção das vagens foi extraído individualmente através do método Trizol (Invitrogen®), seguido de reações de PCR qualitativo utilizando iniciadores específicos para um fragmento de 206pb, para avaliação da presença do dsRNA no tecido vegetal. Para cada reação foram utilizados 100 ng de RNA total.
Através das análises por PCR, foi observado a presença do dsRNA e sua estabilidade e integridade após 24h (Figura 1), sendo principalmente detectado nas secções da vagem próximo ao pedúnculo, local onde foi administrado o dsRNA. A estabilidade do dsRNA 24 horas após a aplicação no tecido vegetal evidenciando o potencial de aplicação dessa tecnologia tanto para ensaios de validação de genes em insetos pragas da soja e/ou seu uso potencial como ferramenta de controle.
Dada a variação no perfil de translocação entre as 5 vagens avaliadas, sugere-se que mais testes sejam conduzidos, tanto variando a dose como tempo de incubação. Adicionalmente, ensaios com manutenção do fluxo hídrico, o que pode favorecer a translocação do dsRNA, podem ser considerados.
Joan Brigo Fernandes, Engenheiro Agrônomo, Doutorando em Agronomia Fitossanidade, UEL

Receba por e-mail as últimas notícias sobre agricultura